|
Les
Picornaviridae
|
1. Morphologie
Le nom de la famille vient de « pico » (petit) et rna (à ARN). Ce sont donc des petits virus à ARN.
Ils sont nus à capside icosaédrique contenant un ARN monocaténaire (+) directement codant. Son diamètre est de 24-30nm. C’est une des familles virale les plus diverses, on compte plus de 200 sérotypes. La poliomyélite fut découverte en 1909 puis isolée en 1930.
Les sous-familles sont classées selon la dissémination sérologique, l’influence du pH…C’est :
- Aphtovirus (muqueuses mammaires et buccales).
- Cardiovirus (cellules du cœur).
- Entérovirus (du tractus digestif).
- Hepatovirus.
- Rhinovirus (tractus respiratoire).
- Non assignés.
|
Genre
|
Entérovirus
|
Cardiovirus
|
Rhinovirus
|
Aphtovirus
|
| Principaux membres (nombre de sérotypes) |
Poliovirus (3),Echovirus
(32), Coxsakievirus (29), Hépatite A (1)
|
Encéphalomyocardie (1)
|
Rhinovirus humain (>100)
|
Fièvre aphteuse (7)
|
| Hôtes principaux |
Hommes et vertébrés
|
Hommes, rongeurs et vertébrés
|
Homme
|
Ongulés artiodactyles
|
| Habitat principal |
Tractus digestif et SNC
|
Cœur et SNC
|
Tractus respiratoire
|
Infection géneralisée
|
| Maladie caractéristique |
Diarrhée, paralysie
|
Myocardite
|
Rhume
|
Eruptions vésiculaires (bouche
et extrémités des membres)
|
| Coéfficient de sédimentation |
155S
|
155S
|
155S
|
145S
|
| Densité en CsCl (g/ml) |
1,34
|
1,34
|
1,39-1,42
|
1,43-1,45
|
| Sensibilité au pH |
Stable à pH 3 à 9
|
Stable à pH 3 à 9
|
Labile à pH<6
|
Labile à pH<7
|
C’est un virus qui a beaucoup évolué, il peut être regroupé en 3 grandes catégories.
2. Génome
Le génome est composé d’un petit ARN (7.2 à 8.5 Kb) simple chaîne (+) codant directement. La structure générale est conservée à travers tous les picornavirus.
L’organisation du génome se fait en 2 parties :
VPg, en 5’, est une protéine, de 23 acides aminés, liée au 1er nucléotide (Viral Protein genomic). La structure va être particulière ce qui permet une régulation de la traduction. L’ARN est directement infectieux lui-même mais moins que le virus entier. Si on introduit l’ARNv dans le milieu, on a production virale avec des déchets. Par transfection, on atteint 80-90% d’infectivité caractéristique des ARN (+).
L’UTR5’ représente 600-1200 nucléotides. Il serait important pour la traduction mais aussi l’encapsidation.
L’UTR3’ (50-100 nucléotides) servirait dans la synthèse du brin d’ARN (-).
Il y a expression d’un polypeptide précurseur de 2100-2400 acides aminés sous clivé ensuite.
Pour les Cardiovirus et Aphtovirus, la petite séquence entre VPg et UTR5’ est un polyC. La longueur de celui-ci varie de 50 à 100 nucléotides. Pour les autres virus, le taux de polyC est plus faible.
3. Cycle de réplication
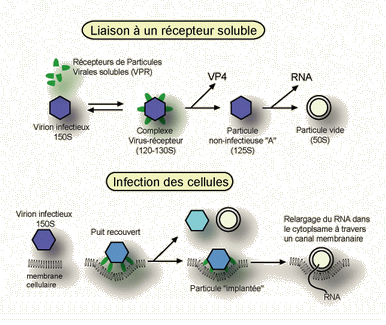
Les récepteurs des picornavirus ont facilement été identifiés.
On peut avoir des récepteurs spécifiques ou des molécules Ig Like (autres molécules : intégrine like). Les capsides sont très immunogènes, ce sont des virus avec espoirs de vaccins (récepteurs en cours d’étude).
La pénétration des virus se fait par endocytose, le déshabillage a lieu dans les vésicules et commence par la perte de VP4, suivi de la traduction (synthèse de P1, P2, P3…). Parallèlement, dans le noyau, on a réplication en 2 temps, synthèse d’un brin (-) qui servira de matrice.
Les ARN (+) migrent dans le cytoplasme pour être empaquetés dans des précapsides qui seront maturées après entrée dans le génome.
On prend des récepteurs des particules solubles et des virions. Un complexe se forme. Naturellement, un déshabillage va avoir lieu, une déstructuration a lieu (passe de 120-130S à 125S). Cela se traduit par le départ de VP4. La particule devient non infectieuse. L’ARN part, on récupère des capsides vides.
Ex vivo : On part d’un virion infectieux mis au contact avec une membrane. On a formation d’un puit recouvert. Soit on obtient des particules vides, soit la particule s’implante. Quand elle s’implante, VP4 part et on a libération de l’ARN.
|
Temps après l’infection |
Evènement |
|
1-2h |
Diminution des synthèses cellulaires ; Condensation de la chromatine (perte de l’apparence homogène du noyau). |
|
2.5-3h |
Début des synthèses virales |
|
3-4h |
Perméabilisation de la membrane plasmique. |
|
4-6h |
Assemblage du virus dans le cytoplasme (les cristaux deviennent parfois visibles). |
|
6-10h |
Lyse cellulaire, relargage des particules virales. |
La traduction du génome viral a pu être notée par marquage 30 mn après infection. Des protéines virales sont synthétisées pour bloquer les synthèses cellulaires (Shut off). Le virus produit une CBP (Cap Binding Protein) se liant aux messagers cellulaires (un précurseur de 220 kD va être clivé et bloquer l’expression des messagers cellulaires).
Il y a synthèse d’un polypeptide géant qui va être clivé en 3 domaines P1, P2, P3 (247 kD chez le poliovirus). Ce clivage se fait en P1 et P2-P3 par une protéase présente dans la capside : la 2A (très spécifique). Les autres sous clivages se font par une autre protéase : la 3C. La 3D est la polymérase, 3B est la protéine VPg, P1 va coder pour des protéines de structures (capside), P1 sera clivé en VP1, VP3 et VP0. VP0 est ensuite clivé en VP2 et VP4. Tant que le clivage n’est pas fait, le virion n’est pas infectieux. Des fragments peuvent avoir des activités de sous clivage.
 VP4
est ici représenté en jaune
VP4
est ici représenté en jaune
3.1. Réplication du génome
La réplication se fait par la protéine polymérase codée par le virus : la 3D
La 3D synthétise le brin (-) en commençant par la queue poly A (VPg-polyU sert d’amorce) ou alors, un facteur intervient induisant une courbure. Le brin (+) et sa queue polyA sert d’amorce.
3.2. Assemblage
3.3. Structure en canyon
|
La capside contient la serrure. La protéine de surface du virus possède la structure en canyon. Le récepteur spécifique (PVR par exemple) permet la reconnaissance de la capside. Avec des anticorps monoclonaux, il reste une infectivité résiduelle, d’autres protéines peuvent alors intervenir. Cette structure est portée par VP4. L’externalisation a lieu au moment du clivage VP2-VP4.
|
4. Infectivité – pathogénicité
Malgré un grand nombre de sérotypes, ceux-ci varient peu d’un type à l’autre.
4.1. Poliovirus
L’infection se fait par ingestion (terre souillée, cultures non lavées…) le cycle est donc oro-fécal.
4.2. Coxsackie
Il vient d’une région américaine, de souris en 1948. Deux types : groupe A et B.
Le virus A (CAV) : provoque des inflammations musculaires très aiguës et des nécroses. (11 sérotypes).
Le virus B (CBV) : (6 sérotypes), ce sont des maladies dégénératives, elles peuvent causer des plaques dégénératives au niveau du pancréas.
Les virus B servent de modèles chez la souris pour étudier le diabète.
L’infection par Coxsackie chez l’homme est souvent associée à des bactéries (paralysies, méningites) et mener à des infections subcliniques. Dans le cas où il donne des myocardites, il n’y a pas de traitements efficaces.
4.3. Echovirus
« Echo » pour Enteric Cytopatic Human Orphan littéralement maladie orpheline de dégénérescence des cellules intestinales. Il existe environ 10 sérotypes pour les échovirus, 1 sérotype pour les rhéovirus.
Ils donnent des maladies intestinales le plus souvent. Depuis 1970, on donne des numéros aux virus.
|
Type |
Maladie |
|
68 |
Pneumonie |
|
69 |
Aucune |
|
70 |
Conjonctivite hémorragique aiguë |
|
71 |
Méningite |
|
72 |
Virus de l’hépatite A |
4.4. Rhinovirus
C’est le virus le plus répandu, il est fragile et donne des froids (T°opt 33°C) et sont facilement éliminés avec la fièvre.
Ils atteignent les enfants, personnes âgées et immunodéprimées.
4.5. Aphtovirus
Ils causent le FMD (Foot Mouth Disease). Eradiqué jusque là (UK, Amérique du Sud…), il est reparti dernièrement. Malheureusement, cela coûte moins cher d’abattre un troupeau que de combattre le virus, la maladie perdure toujours.
Les aphtovirus sont très sensibles à l’acidité. Comprenant 7 sérotypes, ce sont eux qui possèdent les plus gros génomes (8 Kb). La région polyC fait 100-170 nucléotides.
4.6. Cardiovirus
Un seul sérotype, il regroupe l’EMCV (EncephaloMyoCardio Virus).
L’EMCV, le mengovirus dériveraient tous d’un même virus ou alors ils ont des propriétés différentes.
Le génome est d’environ 7.8 Kb. La région non traduite en 5’
contient un polyC de 100-170 nucléotides, comme chez les aphtovirus.
__________________________________