|
Rétrovirus
: Généralités
|
Les Retroviridae sont composés de 3 familles : Les Oncornavirus, Lentivirus et Spumavirus.
|
LES RÉTROVIRUS
|
||
|
Oncornavirus
|
Lentivirus
|
Spumavirus
|
|
HTLV1 (homme), HTLV2 (homme) et rétrovirus animaux associés )des leucémies ou à des cancers. |
VIH1 (homme), SIV (singe), VIH2 (homme), VISNA (mouton), FIV (Chat)… | Rétrovirus retrouvé dans pratiquement toutes les espèces. |
|
POUVOIR PATHOGÈNE
|
||
|
Leucémie et cancers.
|
Virus associés à des maladies à évolution lente accompagnées de troubles neurologiques ou pulmonaires. |
Sans rôle pathogène connu.
|
|
PROPRIÉTÉS BIOLOGIQUES
|
||
| Virus Transformants : (virus succeptibles d'être responsables d'une transformation viro-induite, c'est-à-dire du passage de cellules saines en cellules malignes). | Virus Cytopathogènes :(virus qui détruisent les cellules qu'ils infectent). | |
Les Oncornavirus ont de nombreux virus associés à des mammifères (HTLV, MMV).
Les Lentivirus comportent le VIH1 et VIH2 ainsi que des virus associés (SIV, VISNA, CAEV…). Le VISNA chez le mouton est important car ses symptômes sont similaires à ceux de la sclérose en plaque d’où un intérêt pour le HIV et la sclérose en plaque dans la recherche.
Les Spumavirus sont appelés ainsi car lorsqu’ils détruisent les cellules, une mousse est produite.
Le MPMV est important car est un virus lentivirus simien classé maintenant dans les oncornavirus (par ses propriétés) induisant des transformations. Dans son génome, il n’y a pas de séquences de recombinaison.
Les rétrovirus sont devenus importants depuis 1981 mais étaient présents bien avant…
● 1904 : Ellerman et Bang, cherchant une cause infectieuse pour la leucémie, ont étudié la leucémie chez la poule et réussirent à transférer la maladie d’une être à un autre via un filtrat de tissu.
● 1905 : Paul Erlich propose la théorie sur la surveillance immunitaire. Les cellules tumorales émergeaient spontanément dans l’organisme mais détruit par le système immunitaire.
● 1911 : Peyton Rous réussit à transmettre des tumeurs solides de poulet en transplantant le tissu. Il a isolé l’agent infectieux en plus.
●1960 : Howard Temin savait que le génome du rétrovirus était composé d’ARN et observa que la réplication était inhibée par l’actinomycine D (inhibe la synthèse d’ADN si bien qu’il proposa le concept de la reverse transcription (Prix Nobel pour Baltimore et Temin en 1975).
●1969 : Huebner et Todaro proposèrent l’hypothèse oncogène virale. Les virus peuvent transmettre le cancer car en sont porteurs.
●1970 : la guerre au cancer de Richard Nixon (projet post Kennedy) ne réussi pas à trouver un agent rétroviral qui causait le cancer.
●1981 : Découverte du HTLV1 (Human T cell Leukaemia Virus 1).
●1982 : Découverte du HTLV2.
●1983 : Découverte du LAV (Lymphe Adenopathy Virus) ce sera le HIV1.La grande partie des rétrovirus infectent les vertébrés. On pense que certains pourraient infecter les vertébrés.
●1984 : Découverte du HIV2.
|
Genus:
|
Type Species:
|
|
Avian
type C retroviruses
|
avian
leukosis virus (ALV)
|
|
BLV-HTLV
retroviruses
|
bovine
leukaemia virus (BLV)
|
|
Lentivirus
|
human
immunodeficiency virus (HIV-1)
|
|
Mammalian
type B retroviruses
|
mouse
mammary tumor virus (MMTV)
|
|
Mammalian
type C retroviruses
|
murine
leukaemia virus (MLV)
|
|
Spumavirus
|
human
spumavirus (HSRV)
|
|
Type
D retroviruses
|
Mason-Pfizer
monkey virus (MPMV)
|
Historiquement, les rétrovirus étaient divisés en groupes basés sur leur morphologie en microscopie électronique :
● Type A : Particules internes (intracisternique) non enveloppées. On ne sait pas si elles sont infectieuses, elles sont immatures, observables uniquement à l’intérieur des cellules. Elles proviendraient de rétrovirus ou de séquences rétrovirales partielles endogènes.
● Type B : Elles sont enveloppées, extracellulaires avec un core excentré. Elles contiennent des spicules très proéminentes.
● Type C : Les mêmes que les B sauf que le core est central. Les spicules sont soit vraiment petites voire invisibles. C’est ce qui rassemble le plus grand nombre de virus aviaires et mammifères.
● Type D : Légèrement plus grands que les C (120 nm). Les spicules sont encore plus petites (ex : Mason Pfizer Monkey Virus (MPMV)).
1. Structure
C et D sont assez proches morphologiquement mais ont des propriétés différentes. D est moins immunodépressive mais sont plus transformantes.
Ces particules (A,B,C et D) ont une structure semblable.
Les protéines sont :
● SU : Glycoprotéine de surface. C’est un antigène viral majeur. C’est celle qui est responsable de la fixation sur les récepteurs spécifiques. Elle est souvent associée à la protéine TM (TransMembranaire).
● TM : Elle traverse l’enveloppe virale et
correspond à la partie interne de SU.
● Enveloppe lipidique : contient les protéines virales et cellulaires.
● MA (Matrix Protein): C’est celle qui est la frontière intérieure de l’enveloppe produite par le gène gag.
● CA (Capside) : Elle est aussi codée par gag. Elle protège le core, c’est la protéine la plus abondante.
● NC (Nucléocapside) : Codée par gag. Elle protège le génome. C’est l’assemblage entre ARN et NC avec en plus quelques protéines qui vont former le core.
● PR (Protéase) : Codée par le gène pol. Elle intervient aux 1ers clivages de gag avec certaines protéines de régulation.
● RT : Codée par pol, elle sert à la rétrotranscription en un ADNc qui va ensuite s’intégrer.
● IN (Intégrase) : Codée par pol, elle sert à l’intégration du génome au génome cellulaire.
Il peut y avoir des analogies de séquences entre SU et TM qui permettent de s’associer :

La capside du virus a été difficile à isoler. Le core, non mature, est un peu diffus au départ. Il devient plus dense et central jusqu’à une forme tronconique tout au long du temps, le virus est mature.
2. Le génome
Il est composé de 2 LTR (Long Terminal Repeat) et 3 gènes de base gag, pol, env.
Dans les capsides des rétrovirus, on a des ARN simple brin (+) qui a une coiffe en 5’ et un polyA en 3’.
On trouve 2 molécules dans la capside. La taille des rétrovirus varie entre 8-15kb.
Ces rétrovirus ont 4 caractères principaux :
- Ce sont les seuls virus à être diploïdes véritables (on a 2 chaînes différentes).
- Ce sont les seuls virus à ARN dont le génome est produit par la machinerie cellulaire de transcription (Pas de participation de pol virale en dehors de la RT).
- Ce sont les seuls virus dont le génome va nécessiter un ARN cellulaire spécifique pour la réplication. L’ARN cellulaire est un ARNt.
- Ce sont les seuls virus à ARN (+) qui ne sont pas directement codant, il n’agit pas comme un ARNm.
Les 2 molécules de l’ARN génomiques sont liées par des ponts hydrogène. Il y a d’autres types d’acides nucléiques présents dans les particules : ADN cellulaire, ARNt (tARNPro, tARNLys, tARNTrp) qui sert d’amorce.
Structure du génome
● R : petite région variant de 18-250 nt formant une région unique répétée à chacune des extrémités.
● U5 : Séquence unique (75-250nt). C’est la 1ère partie du génome viral qui va être rétrotranscrite (fera la partie 3’ de l’ADNc).
● PBS : Primer Binding Site, cette séquence correspond à 18 nt qui sont complémentaires de l’extrémité 3’ de l’ARNt qui sert d’amorce.
● Leader (90-500nt) : c’est une région non traduite présente à toues les extrémités 5’ des virus. Elle comporte plusieurs séquences de régulation. Elle s’arrête au début du gène gag. Elle contient la TATA box.
● Les gènes : Ce sont gag, pol et env.
● PPT (PolyPurine Tract) : Cette région riche en A et G comporte 10-12 nt. Elle est responsable de l’initialisation du brin (+) pendant la transcription inverse. Dans le cas des Lentivirus, on a 2 PPT, une centrale et une terminale.
● U3 : région unique non codante (200-1200nt). Elle formera l’extrémité 5’ de l’ADNc et contient la région des promoteurs avec des régulations.
3. Réplication
La reconnaissance du récepteur se fait par SU. La spécificité différencie 3 types de virus :
- Virus Ecotropic : infectent seulement les cellules de souris.
- Virus Xenotropic : infectent toutes les cellules de rongeurs autres que celles de souris.
- Virus Amphotropic : infectent toutes les cellules de rongeurs.
On peut avoir des virus endogènes et exogènes qui utilisent le même récepteur. On détermine des groupes d’interférence pour les virus.
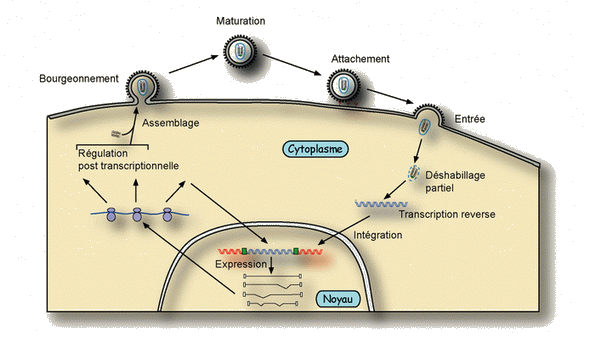
Schéma de la réplication :
Il est probable que le la liaison au récepteur résulte d’un changement conformationnel de la spicule glycoprotéique révélant ainsi le domaine de fusion de TM jusque là masqué et provoquant la fusion de l’enveloppe virale avec la membrane cellulaire.
La pénétration et le déshabillage sont peu compris, mais maintenant on sait que le déshabillage est seulement partiel libérant ainsi le core (nucléocapside) dans le cytoplasme. La transcription reverse a lieu dans la structure organisée de ce core avec des réactants (RT+ ARN+ Nucléotides) libres en solution, la transcription reverse est initiée mais ne peut être complétée et avorte juste après.
4. La transcription reverse
L’ADNc double brin ainsi obtenu (appelé aussi provirus quand il est intégré) va s’intégrer dans l’ADN cellulaire.
Les formes réplicatives auront soit 2 LTR aux extrémités, soit un LTR pour la forme circulaire.
5. Intégration
Elle se fait grâce à l’intégrase (enzyme très spécifique). Il y a un hasard au niveau du génome cellulaire (alors que le virus est spécifique), l’intégration se fait totalement au hasard. Des points chauds de recombinaison pourraient l’être aussi pour l’intégration composée peut-être de A et de T alors que c’est riche en G et C.
L’IS serait doublé et servirait de site d’intégration. Ces séquences varient de 2 à 8 nt. 50% de la séquence supplémentaire disparaît. Au moment de l’excision, il peut y avoir coupure nette ou pas (récupération de nt cellulaires).
Si on enlève des nucléotides des extrémités U3 et U5, on risque d’ôter la mobilité à la structure et donc perdre l’évolution possible du virus.
6. Expression du génome
Les rétrovirus vont utiliser la machine transcriptionnelle cellulaire. Ces virus utilisent des protéines de régulation d’origine virale et réguler le virus mais aussi le système cellulaire. Le génome viral fonctionne à peu près comme de l’ADN cellulaire puisqu’il est intégré. Il dépend donc de l’état de la cellule ce qui créé une interaction entre la cellule et le virus.
Le maximum d’expression est obtenu par divers moyens : chevauchement, déplacement des cadres de lecture, synthèse de précurseurs qui seront clivés.
U3 contient la région promoteur et une TATA box. Ces LTR ont été séquencés et beaucoup analysés. Les séquences NFКB et SP1 vont être régulée par des protéines cellulaires.
6.1 Les épissages
Ex : Chez l’ALV et le MLV, un précurseur (transcrit primaire) correspond aux gènes gag, pro, pol. Quand il est épissé, on obtient env.
Cet épissage est régulé par l’appareil cellulaire en cis (sur les cis séquences).
6.2 Expression différentielle
Elle se fait par saut de ribosome.
|
Pour le HIV, pro chevauche gag et pol. Deux séquences vont être exprimées : gag-pro-pol et gag seul. Quand le ribosome finit gag, il revient en arrière pour faire pro et pol. |
|
|
Pour le HTLV, on obtient gag, gag-pro et gag-pro-pol. Pro va cliver gag-pro et gag-pro-pol. Le ribosome revient en arrière pour synthétiser pro-pol. |
|
| Pour le MLV, gag est synthétisé (UAG= terminaison). Par épissage, on a gag-pro-pol avec perte de l’UAG. On a ensuite une coupure pour obtenir pro et pol. |
7. Assemblage
Pour les particules B et D, l’assemblage a lieu dans le cytoplasme comme pour d’autres virus.
Pour les virus de type C (ex : HIV), l’assemblage se fait tout près de la surface cellulaire puis bourgeonnement, relargage et maturation.
Dans tous les cas, la maturation a lieu après sa sortie.
8. Vecteurs rétroviraux
Une séquence va intervenir, c’est la séquence ψ ou séquence d’empaquetage. On construit un vecteur LTR-ψ-ADNc-LTR pour voir si l’ADNc est inséré.
On peut faire une séquence LTR-ψ-néomycine-promoteur-ADNc-LTR. Pour voir l’expression de la protéine. Les séquences ψ sont très dangereuses à manipuler.
___________________________________________